НИР лаборатории направлены на решение, как фундаментальных проблем, так и на социально ориентированные цели и задачи. В фундаментальной области – важнейшими в общей биологии являются проблемы биологического вида, его дефиниции и идентификации. Вопросы видообразования, филогенеза и соответствующих эволюционных реконструкций, немыслимы сейчас без конструирования молекулярных филогений. Общепринята в настоящее время синтетическая теория эволюции (СТЭ), частью которой является биологическая концепция вида (БКВ). Обоснование и систематическое изложение СТЭ представлено в работах Холдейна (Haldane, 1932), Добжанского (Dobzhansky, 1937; 1943; 1951; 1970), Хаксли (Huxley, 1954), Майра (Майр, 1968; Mayr, 1982). В России популярным обобщением СТЭ стала книга Тимофеева-Ресовского и соавторов (1977). Конструктивное развитие положений СТЭ и БКВ предложено, Воронцовым (1980), Харрисоном (Harrison, 1998) и др.
Одно из уязвимых мест СТЭ – отсутствие, как правило, возможности экспериментальной проверки ключевого в БКВ критерия не скрещиваемости видов в природе. Недавно, с развитием филогенетики, возникли новые противоречия между БКВ и филогенетической концепцией вида (Avise, Wollenberg, 1997; Avise, 2000; Harrison, 1998; Templeton, 1998), которые доходят даже до отвержения значимости для филогенетики текущих микроэволюционных процессов. Не вполне разработана в СТЭ и теория видообразования. Точнее говоря, в количественно-аналитическом смысле теория как таковая вообще не сформулирована, а имеется только некоторый набор концепций. Решение этих вопросов требует переописания биоразнообразия на современной молекулярной и биоинформационной основе, включая радикальное обновление музейных коллекций с формированием и пополнением БД общедоступных через Интернет.
В качестве обобщения в этом направлении можно сослаться на одно из основных достижений лаборатории за 2017 г.
Предложен оригинальный подход, базирующийся на специальной программной функции BIN (Barcoding Index Number) получения кластеров нуклеотидных последовательностей по молекулярным маркерам (ММ), и их сравнения с традиционной таксономической классификацией. Используя BIN и массив международной базы данных BOLD впервые удалось осуществить идентификацию таксонов рыб трех рангов вид, род, семейство и проделать статистическую оценку состоятельности идентификации. По большому массиву данных обнаружено, что ошибки идентификации по ММ не превышают 20%, а возможная ретикуляция генных деревьев достигает не более 16%. На основе полученных данных и материалов о связи молекулярной эволюции с иерархией таксонов в мире животных в целом, обосновывается соответствие молекулярной эволюции и существующих общебиологических и эволюционных парадигм, Нео-Дарвинизма и биологической концепции вида (Картавцев Ю.Ф., ННЦМБ ДВО РАН). Идентификация видов на основе молекулярных маркеров (ММ), а точнее на основе последовательностей нуклеотидов ДНК или ДНК-штрихкодов (ДНК-штрихкодирование, iBOL; www.ibol.org ), является весьма успешной, как убеждают обширные данные мировой базы данных (БД), BOLD (www.bold.org). Однако теория, которая объяснила бы данный факт, пока не создана. В представляемой работе предложен оригинальный подход, базирующийся на Величине Индекса ДНК-штрихкодирования (Barcoding Index Number, BIN). Используя BIN и массив БД BOLD удалось осуществить идентификацию (выделение) таксонов трех рангов (вид, род и семейство) и проделать статистическую оценку состоятельности идентификации, которая достигает более 80% (Рис. 1). Кроме того, доказано, что для большинства построенных генных деревьев для маркера Со-1 (ген цитохром оксидазы с, субъединица 1) существует хорошее соответствие их топологии и общепринятой таксономической классификации в иерархии таксонов вид, род, семейство, по меньшей мере для проанализированных обширных данных по рыбам; коэффициент детерминации связи составил: (R2 = 0.84–0.98, P < 0.001; Рис. 2). Полученные знания об отклонениях от предсказанных значений для идентификации видов и определения ранга других таксонов позволят улучшить систему описания биоразнообразия на точной молекулярной и биоинформационной основе. Основной вывод из проделанного анализа состоит в том, что успешная идентификация по ММ возникает вследствие преобладания в природе географического способа видообразования. В ходе длительного во времени процесса формирования видов, с накоплением мутаций и соответствующих многочисленных нуклеотидных замен в различных дочерних предковых таксонах, могут возникать уникальные последовательности нуклеотидов, ДНК-штрихкоды, которые обнаруживаются посредством ММ. Последующая независимая эволюции возникших филетических линий, включая дальнейшую дивергенцию до уровня рода и семейства и так далее, и составляет фактическую базу обнаруженной связи величины значения BIN и ранга для трех таксонов, выделенного посредством традиционного морфологического подхода (Рис. 2). В целом связь генетических расстояний и ранга таксона доказана для различных групп животных по данным для отдельных генов и для полных митогеномов (Рис. 3).
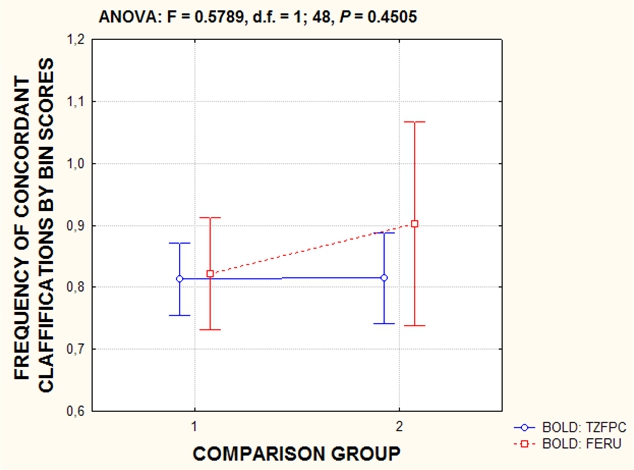
Рис. 1. Двухфакторный дисперсионный анализ распределения средних значений классификаций на основе BIN в двух исследованиях рыб, суммированных по базе данных BOLD.
Средние значения для BIN показывают согласованные классификации (%, Y-ось) по молекулярному маркеру Co-1 мтДНК видов/образцов с независимым диагнозом по традиционным признакам, представленным в BOLD. Группы сравнения (Comparison group, X-ось) представлены двумя категориями: отдельные виды (1) и самостоятельные роды (2). Значения согласованной классификации (concordant classifications) не различаются в двух группах (1 и 2). Оценки также не отличаются между двумя исследованиями (Steinke et al., 2009, TZFPC; Turanov et al., 2014, FERU), которые представили 1219 и 285, а также 199 и. 64 проанализированных образцов для видов и родов в двух цитированных работах. Суммарная согласованность классификаций на основе оценок BIN составила более 82% (см. точки с доверительными интервалами на графике); соответственно величина ошибочных классификаций составляет менее 18% (по Kartavtsev, 2017).
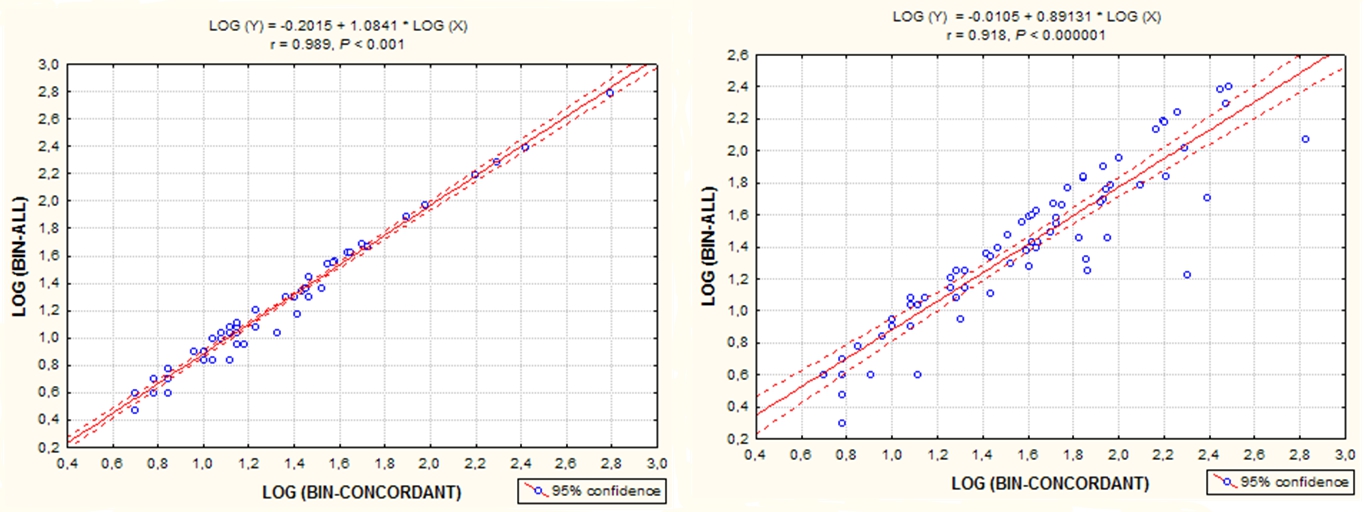
Рис. 2. Регрессионный анализ распределения значений BIN, размещенных в базе данных BOLD для трех проектов исследования рыб, FERU/TZFPC и SCFAA (По Kartavtsev, 2017).
Значения по Y-оси, LOG (BIN-ALL) (переменная, представляющая значения зоологически определяемых образцов для трех категорий, обозначенных в BOLD: внутри видов, внутри родов и внутри семейств) в сопоставлении со значениями по X-оси, LOG (BIN-CONCRDANT) (Согласующиеся OTU-кластеры для Co-1 мтДНК ДНК-штрихкодов или образцов последовательностей для тех же трех выше описанных категорий). Изменчивость показывает статистически значимую положительную линейную зависимость двух переменных X и Y для проанализированных проектов FERU/TZFPC (слева; Steinke et al., 2009, TZFPC; Turanov et al., 2014, FERU) и SCFAA (справа; McCusker et al. 2012). Суммарная ковариация двух переменных на основе значений BIN в проектах составила: R2 = 98% и R2 = 84%, соответственно для двух графиков. То есть, ошибки в детерминации составили: 2-16%. Подробности представлены в статье (Kartavtsev, 2017; Supplement, DATA1, DATA2, DATA3 spreadsheets).
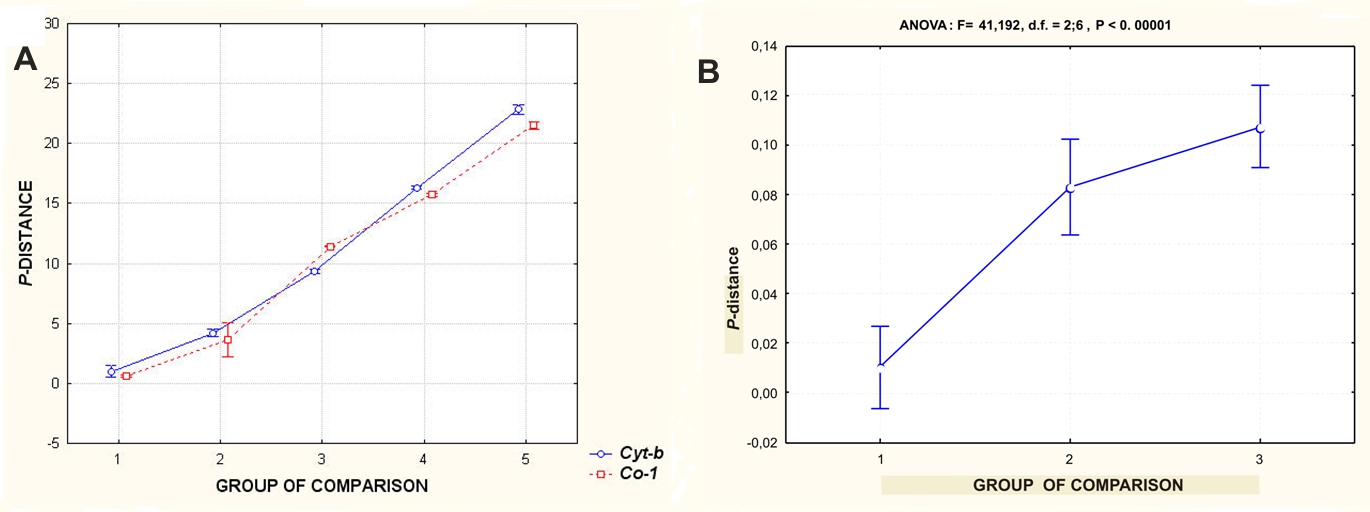
Рис. 3. Распределение р-расстояний в таксонах животных.
График (А) слева демонстрирует изменчивость средних значений р-расстояний (P-distance; ось-Y) для последовательностей генов Cyt-b и Co-1 различных таксонов животных в зависимости от ранга групп, в двухфакторном дисперсионном комплексе. Группа сравнения (Group of Comparison): 1 – внутри видов, между особями одного вида, 2 – внутри подвидов, полувидов и близнецовых видов, между особями этих таксонов, 3 – внутри рода, между морфологически различающимися видами одного рода, 4 – внутри семейства, между видами различных родов одного семейства, 5 – внутри отряда, между видами различных семейств одного отряда. На график отражена изменчивость групп сравнения без взвешивания расстояний на число видов (n). Проиллюстрирован эффект взаимодействия, то есть, различия величин расстояний для двух генов в пяти группах сравнения: F = 101.05, d.f. = 4, 22227, P < 0.0001. Вертикальные линии – доверительные интервалы (95%) для средних значений (По Kartavtsev, 2011a,b). На графике (B) справа показана изменчивость 3 групп сравнения для митогенома, включающего 13 белок-кодирующих генов у ельцовых рыб (Kartavtsev et al., 2017). Для ельцовых рыб включены 3 группы сравнения (Group of Comparison): (1) внутри вида, (2) внутри рода, (3) внутри подсемейства.
Публикации:
Kartavtsev Y. Barcode index number, taxonomic rank and modes of speciation -- examples from fish. MITOCHONDRIAL DNA PART A, 2017. E-publ. doi: 10.1080/24701394.2017.1315570.
Kartavtsev Yu. Ph., Batishcheva N.М., Bogutskaya N.G., Katugina A.O., Hanzawa N. Molecular Systematics Research, DNA Barcoding of Altai Osmans, Oreoleuciscus (Pisces, Cyprinidae, Leuciscinae), and Nearest Relatives, Inferred from Sequences of Cytochrome b (Cyt-b), Cytochrome Oxidase c (Co-1), and Complete Mitochondrial Genome // MITOCHONDRIAL DNA PART A, 2017. V. 28. P.502-517.
Информацию подготовил г.н.с. Картавцев Ю.Ф.










